Science en action 9 avril 2024
- Accueil
- Espace presse
- Communiqués de presse
- Vers un diagnostic itinérant et instantané des maladies virales
Vers un diagnostic itinérant et instantané des maladies virales

Une nouvelle technique va permettre de détecter directement au champ les maladies virales. Ici, champ d'ignames en Guadeloupe © D. Filloux, Cirad
Une nouvelle technique de séquençage à haut-débit miniaturisée et portable* se développe depuis quelques années en santé humaine et animale. Au travers de laboratoires mobiles, cette approche permet de séquencer directement sur le terrain et de façon quasi-instantanée les virus, comme Ebola ou encore Zika. Un diagnostic rapide et précoce, qui permet aussi d’éviter le mouvement d’échantillons contaminés.
« Cette technologie se caractérise par la production de longues séquences nucléotidiques ce qui permet de séquencer intégralement les génomes viraux », explique Philippe Roumagnac, virologiste au Cirad. Le Cirad a été un des tous premiers laboratoires à l’international à tester et à valider son utilisation en virologie des plantes. « A partir d’une plante d’igname malade, nous avons séquencé, en quelques heures seulement, la totalité des génomes de deux virus à ARN simple brin, un macluravirus et un potyvirus » , ajoute son collègue Denis Filloux.
Vers un diagnostic itinérant et instantané des virus des plantes, en appui aux réseaux d’épidémiosurveillance
La validation en laboratoire de virologie végétale de cette approche ouvre la voie, comme en virologie humaine, à la détection sur le terrain, délocalisée, itinérante, et en temps réel des virus des plantes, qu’ils soient chroniques, saisonniers ou émergents. En limitant le temps entre l’échantillonnage de plants malades et les résultats du diagnostic, cette avancée technologique est amenée à aider les réseaux d’épidémiosurveillance à détecter de manière plus précoce la présence d’organismes nuisibles.
Ces travaux, développés par une équipe internationale associant le Cirad à des partenaires européen, indien et sud-africain, ont été financés par la fondation Agropolis dans le cadre du projet étendard E-SPACE (Améliorer l’épidémiosurveillance des maladies des plantes tropicales et méditerranéennes).
* Oxford Nanopore MinION
Référence
Filloux D., Fernandez E., Loire E., Claude L., Galzi S., Candresse T., Winter S., Jeeva ML., Makeshkumar T., Martin D.P., Roumagnac P., 2018. Nanopore-based detection and characterization of yam viruses. Nature - Scientific Reports 8:17879
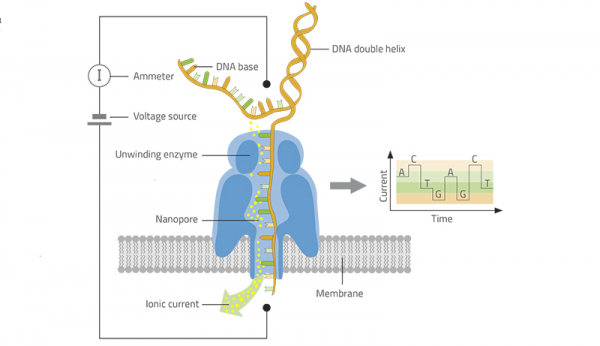
Le séquençage par nanopores, comment ça marche ?
La molécule d’ADN à séquencer est captée par une enzyme de déroulement qui extrude l’ADN simple brin à une vitesse réduite au travers du nanopore. Lors de son passage, l’ADN simple brin est ralenti par la constriction du nanopore. Cela permet la lecture des bases nucléotidiques par mesure de l’intensité du courant électrique traversant le nanopore à un instant donné.
Illustration © Kerstin Göpfrich